Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Handleiding
- Type
- Handleiding

uPath PD-L1 (SP263)-beeldanalyse
voor niet-kleincellige longkanker
algoritmegids


Inhoudsopgave
Inleiding 1
Algoritmesamenvatting en -uitleg 2
Beoogd gebruik 3
Beoogd gebruik van het product 3
Doel van de Algoritmegids 3
Klinische significantie 4
Testprincipes 5
Beperkingen 6
Gegevensbeveiliging 7
Werkstroom voor het gebruiken van de uPath PD-L1-
beeldanalyseapplicatie voor het NSCLC-algoritme 8
Werkstroom van de patholoog 9
PD-L1-beeldanalyse 12
PD-L1-evaluatie 15
Kleuringsevaluatie van het PD-L1-algoritme 15
Kleuringskenmerken 15
Prestatiekenmerken 26
Vergelijking van methoden 26
Reproduceerbaarheidsonderzoeken voor pathologen 27
Reproduceerbaarheidsonderzoeken voor scanners 28
Problemen oplossen 29
Bronvermeldingen 33


Inleiding
De Roche uPath enterprise software (uPath enterprise software)
met de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
algoritme voor niet-kleincellige longkanker (NSCLC) (uPath PD-L1
(SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme)
is een softwaresysteem dat is bedoeld voor ondersteuning
bij de kwantitatieve beoordeling van eiwitexpressie in
immunohistochemisch (IHC) gekleurde histologische coupes van
met formaline gefixeerde, in paraffine ingebedde (FFPE) normale
en neoplastische weefsels.
De uPath enterprise software is een end-to-end-
softwareoplossing voor digitale pathologie waarmee
pathologische laboratoria digitale beelden van pathologische
monsters kunnen verkrijgen, beheren, bekijken, analyseren,
delen met anderen, en erover kunnen rapporteren. Met de
uPath enterprise software kan de patholoog digitale beelden bij
verschillende vergrotingen bekijken, aantekeningen toevoegen,
metingen doen aan weefselcoupes, beeldanalyse uitvoeren, en
rapporten opstellen.
Opmerking: de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor
het NSCLC-algoritme is een toegevoegde computerondersteunde
methodologie ter ondersteuning bij het verkrijgen en meten van
beelden van objectglaasjes met weefselmonster die IHC zijn
gekleurd voor de aanwezigheid van het PD-L1-eiwit. Het is de
verantwoordelijkheid van de patholoog om overeenkomst
te verifiëren door geschikte controles te gebruiken om de
geldigheid van de beeldanalysescores te bevestigen, zoals is
gespecificeerd in het methodeblad van het VENTANA PD-L1
(SP263) Rabbit Monoclonal Primary Antibody (beschikbaar op
www.ventana.com).
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 1

2 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids
Algoritmesamenvatting en -uitleg
Bij de beeldanalyseapplicaties kan de patholoog met de
uPath enterprise software één of meer interessegebieden
(ROI's) selecteren en afbakenen. Elk ROI kan bij
verschillende vergrotingen worden bekeken en vervolgens
worden geanalyseerd met de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme. Het totale
aantal doel-TC wordt gegenereerd en tumorcellen (TC) worden
gestratificeerd afhankelijk van of deze gekleurd of niet-gekleurd
zijn. De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme deelt het aantal gekleurde TC door het totale
aantal TC (gekleurd en niet-gekleurd) om de PD-L1
TC-positiviteitsscore te genereren op een schaal van 0-100%.
De uPath PD-L1 (SP263)-beeldanalyse voor het NSCLC-algoritme
kan een score voor een bepaald ROI genereren of een totale
score voor alle geselecteerde ROI's op dat glaasje. Hoewel het
algoritme niet-tumorcellen detecteert als deel van de algehele
analyse, geven de overlay en output alleen de TC weer die zijn
gebruikt bij het berekenen van de TC-positiviteitsscore. De
patholoog kan de score accepteren die door de uPath PD-L1
(SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme is
bepaald of vervangen door een andere score. De patholoog
moet zorgvuldig controleren wat het algoritme heeft aangemerkt
als positieve en negatieve TC en bevestigen dat het algoritme
correct is of deze handmatig vervangen. De uPath PD-L1
(SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme
voert geen onafhankelijke interpretaties van de gegevens uit en
dient daarom te worden beoordeeld door een gekwalificeerd
patholoog in combinatie met histologisch onderzoek, relevante
klinische informatie en gepaste controles. Het is ontworpen en
wordt aangeduid als een hulpmiddel voor de patholoog bij de
beoordeling van PD-L1-expressie bij de 50%.

Beoogd gebruik
Doel van de Algoritmegids
Deze algoritmegids voor de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor NSCLC (algoritmegids ) is bedoeld om:
• achtergrondinformatie te bieden over het beoogde gebruik
van de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme, de testprincipes en -beperkingen;
• de benodigde materialen, IT, gegevensbeveiliging en
netwerkvereisten te definiëren;
• stapsgewijze gebruiksinstructies te geven voor de uPath PD-L1
(SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme;
• foto's te bieden die aangeven hoe de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme moet
worden gebruikt;
• pathologen een hulpmiddel te geven voor eenvoudiger gebruik
van de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme met FFPE NSCLC-coupes die zijn gekleurd
met de VENTANA PD-L1 (SP263) Assay;
• voorbeeldafbeeldingen te bieden van moeilijke gevallen ter
informatie over het gebruik van de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme bij
dergelijke evaluaties;
• prestatiekenmerken weer te geven van de uPath PD-L1
(SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme.
Beoogd gebruik van het product
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme ondersteunen de patholoog bij de detectie
en semi-kwantitatieve meting van PD-L1-eiwit in met formaline
gefixeerd, in paraffine ingebed NSCLC-weefsel. Indien
gebruikt in combinatie met de VENTANA PD-L1 (SP263) Assay
is het geïndiceerd als hulpmiddel voor de identificatie van
patiënten met NSCLC voor behandeling met de therapieën
met de positiviteitsgrenswaarde ≥50% PD-L1 TC volgens de
goedgekeurde therapeutische productlabels.
Opmerking: de uPath PD-L1 (SP263)-beeldanalyseapplicatie
voor NSCLC is een toegevoegde computerondersteunde
methodologie ter ondersteuning bij het verkrijgen en meten
van beelden van objectglaasjes met weefselmonsters die IHC
zijn gekleurd voor de aanwezigheid van het PD-L1-eiwit. Het is
de verantwoordelijkheid van de patholoog om overeenkomst te
verifiëren en geschikte controles te gebruiken om de geldigheid
van de beeldanalysescores te bevestigen, zoals is gespecificeerd
in het methodeblad van het VENTANA PD-L1 (SP263) Assay
(onderdeelnr. 741-4905).
Dit algoritme is bedoeld voor in-vitrodiagnostiek (IVD).
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 3

Klinische significantie
Longkanker is al tientallen jaren de meest voorkomende vorm van
kanker en blijft de belangrijkste oorzaak van kankersterfgevallen
wereldwijd. Naar schatting is longkanker verantwoordelijk voor
12,9% van alle nieuwe gevallen van kanker en jaarlijks de oorzaak
van bijna 1,76 miljoen sterfgevallen wereldwijd, of ongeveer
één op de vijf kankergerelateerde sterfgevallen. NSCLC is de
meest voorkomende vorm van longkanker en maakt ongeveer
85% van alle gevallen van longkanker uit. Bij de meerderheid
van de patiënten met NSCLC bevindt de ziekte zich in een
inoperabel, lokaal gevorderd stadium (IIIB) of gemetastaseerd
stadium (IV). Voor geen van beide bestaan momenteel curatieve
behandelingsmethoden. De 5-jaars relatieve overleving voor de
diagnose NSCLC in een vergevorderd stadium is 4,7%.
Het programmed death-ligand (PD-L1) is een
transmembraaneiwit dat tot expressie komt op T-cellen die
down-regulatie van immuunreacties veroorzaken door zich te
verbinden aan de twee remmende receptoren van programmed
death-1 (PD-1) en B7-1 (CD80). Het binden van PD-L1 aan
PD-1 remt T-celproliferatie, cytokineproductie en cytolytische
activiteit, wat leidt tot de functionele inactivering of uitputting
van T-cellen. PD-L1 bindt zich ook aan CD80 op antigeen-
presenterende cellen en geactiveerde T-cellen die down-regulatie
van de immuunreactie mogelijk maken, waaronder het remmen
van T-celactivatie en cytokineproductie. PD-L1-expressie wordt
waargenomen in immuuncellen en TC. Afwijkende expressie
van PD-L1 in TC wordt gemeld als belemmering voor anti-
tumorimmuniteit, resulterend in immuunevasie. De opkomst
van immunotherapieën die het PD-L1/PD-1-pathway verstoren
hebben geleid tot verbeterde overlevingskansen voor patiënten
met de diagnose NSCLC. De prognose van de behandeling
is echter afhankelijk van het niveau van PD-L1-expressie en
kwantificatie van PD-L1 met immunohistochemische kleuring is
daarom vereist.
Immunohistochemie kan worden gebruikt om specifieke
antigenen te detecteren die aanwezig zijn in weefselmonsters.
Dit maakt het een effectief hulpmiddel voor pathologen bij het
stellen van een diagnose en prognose van carcinomen. De
VENTANA PD-L1 (SP263) Assay is een monoklonaal antilichaam
van konijn, bedoeld voor laboratoriumgebruik bij de semi-
kwantitatieve detectie van het PD-L1-eiwit in FFPE-coupes van
normaal en neoplastisch weefsel. Een voordeel van histologische
weefselpreparaten is dat de morfologie van het weefsel intact
is waardoor het gemakkelijker kan zijn om te beoordelen of
een patiëntmonster al dan niet positief is voor PD-L1. Alle
histologische tests moeten door een specialist op het gebied
van NSCLC-morfologie en/of pathologie worden beoordeeld.
De resultaten moeten worden aangevuld met morfologisch
onderzoek en de juiste controles en de tests moeten worden
gebruikt in combinatie met andere klinische gegevens en
laboratoriumgegevens.
4 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Testprincipes
De uPath enterprise software van de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme maakt gebruik
van beeldanalysetechnieken om een TC-positiviteitsscore te
verkrijgen.
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-
algoritme maakt gebruik van vooraf gedefinieerde parameters
om beelden van weefsel dat is gekleurd met de VENTANA PD-L1
(SP263) Assay te beoordelen.
Stappen van de beeldanalyse:
• Identificatie van witte ruimte en automatische uitsluiting van
analyse.
• Detectie van cellen op het gehele beeld.
• Classificatie van cellen als TC of andere celtypen.
• Identificatie van gekleurde en niet-gekleurde TC.
• Berekening van de TC-positiviteitsscore door het aantal
gekleurde TC te delen door het totale aantal TC volgens het
methodeblad van de VENTANA PD-L1 (SP263) Assay.
Hoe de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor
het NSCLC-algoritme TC identificeert en hoe de score
wordt berekend:
• De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor
het NSCLC-algoritme identificeert TC op basis van kleur,
intensiteit, grootte en morfologische kenmerken.
• Geïdentificeerde TC worden geclassificeerd als gekleurd met
behulp van gedetecteerde membranen en vooraf ingestelde
grenzen in navolging van het methodeblad van de VENTANA
PD-L1 (SP263) Assay.
• Om het percentage gekleurde TC te berekenen, wordt het
aantal gekleurde TC gedeeld door het totale aantal TC volgens
het methodeblad van de VENTANA PD-L1 (SP263) Assay.
Witte ruimtebepaling door de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme:
• Het algoritme sluit witte ruimte automatisch uit. Artefacten
zoals vuil, krassen of inkt worden mogelijk niet automatisch
uitgesloten.
• De gebruiker kan de gebieden beoordelen die door het
algoritme zijn aangemerkt als witte ruimte door een valse
kleurenoverlay te gebruiken, zie 'PD-L1-beeldanalyse: valse
kleurenoverlay'.
De criteria van de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme voor het
genereren van de TC-positiviteitsscore-output:
• De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor
het NSCLC-algoritme meldt een score tot een decimaal,
bijvoorbeeld '4,8%'.
• Het wordt niet aanbevolen de score naar boven of beneden af te
ronden. '4,8%' moet bijvoorbeeld niet worden afgerond naar 5%.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 5

De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-
algoritme is compatibel met de VENTANA PD-L1 (SP263) Assay.
De testresultaten hangen af van de kwaliteit en nauwkeurigheid
van het IHC-objectglaasje waarvan een beeld wordt gemaakt, en
van het verkregen beeld dat geanalyseerd wordt.
Alvorens beelden van objectglaasjes op de uPath enterprise
software te laden voor analyse, moet de patholoog de kleuringsrun
van de VENTANA PD-L1 (SP263) Assay valideren door handmatig
microscopisch onderzoek van de PD-L1-controleglaasjes om te
verifiëren dat de verwachte resultaten worden behaald.
Volg de aanbevelingen van de fabrikant op voor de VENTANA
PD-L1 (SP263) Assay, waaronder alle positieve en negatieve
kwaliteitscontrolematerialen voor iedere kleuringsrun. Als
de controleglaasjes niet aanvaardbaar zijn voor handmatig
microscopisch onderzoek, moeten de weefsels opnieuw worden
gekleurd, totdat de resultaten wel aanvaardbaar zijn.
De patholoog moet de aanbevelingen voor interpretatie van de
VENTANA PD-L1 (SP263) Assay opvolgen.
Raadpleeg het methodeblad van de VENTANA PD-L1 (SP263)
Assay (onderdeelnr. 1014258NL) en de interpretatiegids
(onderdeelnr. 1015317EN) (verkrijgbaar via www.ventana.com).
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme is bedoeld voor gebruik door een gekwalificeerd
patholoog in combinatie met histologisch onderzoek, relevante
klinische informatie en de juiste controles. Het is niet bedoeld
als onafhankelijk hulpmiddel en vereist deskundige menselijke
interventie gedurende het analyseproces.
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-
algoritme kan onjuiste scores genereren als de vastgelegde
beelden abnormaal zijn gekleurd (nucleair, cytoplasmisch, enz.).
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme verwerpt geëlongeerde tumorcelkernen,
ongeacht de vorm van de hele cel. Om deze reden kan het nodig
zijn om tumoren die een groot aantal cellen met een geëlongeerde
kern bevatten, handmatig te beoordelen.
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme is getraind, ontwikkeld en gevalideerd met
NSCLC-weefselmonsters.
Beperkingen
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme is niet getest en de veiligheid en effectiviteit is
niet gevalideerd voor gebruik met een PC vanuit huis.
Het uPath PD-L1 (SP263)-beeldanalysealgoritme is geïndiceerd
als hulpmiddel voor de identificatie van patiënten met NSCLC voor
behandeling met de therapieën met de positiviteitsgrenswaarde
50% PD-L1 TC volgens de goedgekeurde therapeutische
productetiketten.
De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het
NSCLC-algoritme kan cellen verkeerd identificeren door de
aanwezigheid van zwakke cytoplasmische en/of membraneuze
kleuring, sterke immuuncelkleuring die overlapt met
tumorcelkleuring met significante vermengde ontsteking, TC met
zwakke cytoplasmische kleuring en kleuring van niet-doelcellen.
Dit kan leiden tot het onjuist identificeren van TC als niet-TC en
van niet-TC als positieve TC.
Hoewel macrofagen moeten worden uitgesloten van het
analysegebied, is het niet altijd mogelijk om alle macrofagen uit
te sluiten. De score die wordt gegenereerd door de uPath PD-L1
(SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme kan
daarom zijn beïnvloed door de macrofagen in het ROI dat wordt
geanalyseerd. Dit is uiterst belangrijk als een patiëntscore dicht bij
de grenswaarde van 50% ligt.
Cytoplasmatische kleuring is meestal diffuus en in sommige
gevallen is een granulair patroon aanwezig. In zeldzame gevallen
is er sprake van perinucleaire gespikkelde kleuring met variabele
intensiteit. Het totale percentage signaalintensiteiten van
tumormembranen wordt visueel geschat en gebruikt om het
PD-L1-expressieniveau te genereren. Cytoplasmatische kleuring
van tumorcellen wordt buiten beschouwing gelaten voor het
bepalen van PD-L1-expressie. Een negatief controleantilichaam
met hetzelfde isotype wordt gebruikt om de aanwezigheid van
achtergrond in testmonsters te evalueren en een basisniveau van
de kleuringsintensiteit te bepalen.
6 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Gegevensbeveiliging
Kwaadaardige software of ongeoorloofde toegang kan leiden tot
gegevensverlies of onbeschikbaarheid van het instrument.
De volgende aanbevelingen zijn erg belangrijk om een virus door
kwaadaardige software of ongeoorloofde toegang en misbruik
van het instrument te voorkomen:
• Installeer of open geen andere software op het instrument.
• Zorg dat de andere computers en diensten op het netwerk
goed zijn beveiligd tegen kwaadaardige software en
ongeoorloofde toegang. Dit geldt bijvoorbeeld voor het
laboratoriuminformatiesysteem (LIS), het archiefsysteem en
het back-upsysteem.
• Klanten zijn zelf verantwoordelijk voor de beveiliging van het
netwerk in hun werkomgeving, voornamelijk als het gaat om
de bescherming tegen kwaadaardige software en aanvallen.
Deze bescherming kan bestaan uit maatregelen, zoals een
firewall, om het apparaat te scheiden van ongecontroleerde
netwerken. De bescherming kan ook maatregelen bevatten
die ervoor zorgen dat het verbonden netwerk geen
kwaadaardige code bevat.
• Beperk de fysieke toegang tot het instrument en alle
gekoppelde IT-infrastructuur (computers, kabels,
netwerkapparatuur, enz.).
• Zorg dat de back-up- en archiefbestanden van het instrument
zijn beschermd tegen ongeoorloofde toegang en rampen.
Dit geldt ook voor externe opslaglocaties, locaties voor
calamiteitenherstel en de veilige overdracht van back-
upbestanden.
• Gebruik indien mogelijk een firewall om het netwerkverkeer te
beperken.
• USB-sticks kunnen worden gebruikt voor verschillende
typen back-ups en herstelbewerkingen. Onjuist gebruik
van een USB-stick kan leiden tot gegevensverlies of een
apparaatstoring.
• Gebruik alleen USB-sticks die zijn getest en geïnstalleerd door
uw plaatselijke Roche-servicevertegenwoordiger.
• Er kan slechts één USB-apparaat tegelijkertijd worden
gebruikt. Controleer of er geen andere USB-apparaten zijn
verbonden voordat u een USB-stick plaatst.
• Gebruik de knop Uitwerpen in Windows voordat u een
USB-stick verwijdert.
• De standaardconfiguratie van het besturingssysteem (OS) dat
met de server wordt meegeleverd, mag niet worden gewijzigd
omdat dit gevolgen heeft voor de gehardende
OS-configuraties.
• Gebruik de USB-stick uitsluitend op het apparaat om te
voorkomen dat een virus de uPath enterprise software infecteert .
• Sla geen andere gegevens op deze USB-stick op.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 7

Werkstroom voor het gebruiken van de uPath PD-L1-beeldanalyseapplicatie
voor het NSCLC-algoritme
Benodigde materialen
• uPath enterprise software
• uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme
• NSCLC-weefselglaasjes gekleurd met de VENTANA PD-L1 (SP263) Assay (met gebruik van de OptiView DAB IHC Detection Kit)
op het BenchMark ULTRA-instrument
• VENTANA DP 200-objectglaasjesscanner
Werkstroom
1. Een NSCLC-weefselglaasje wordt gekleurd met de VENTANA PD-L1 (SP263) Assay op een BenchMark ULTRA-instrument.
2. Beeldacquisitie (scannen van volledige glaasjes) wordt uitgevoerd met de VENTANA DP 200-objectglaasjesscanner met
20x-vergroting op een z-vlak.
3. Als er eenmaal digitale beelden zijn verkregen, worden deze beelden overgebracht van de computer die is gekoppeld aan de
VENTANA DP 200-objectglaasjesscanner naar het beeldbeheersysteem (IMS) op een gecentraliseerde server.
4. Na de overdracht naar de server wordt er een casus aangemaakt in de uPath enterprise software. Casussen kunnen automatisch
worden aangemaakt door middel van communicatie met het laboratoriuminformatiesysteem (LIS) met behulp van identificatie-
informatie (bijv. weefseltype en primair antilichaam) op het etiket met de streepjescode van het objectglaasje of handmatig ingevoerd
op de uPath enterprise software (raadpleeg de gebruikershandleiding van de uPath enterprise software [onderdeelnr. 1018943NL]).
5. Als de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme is geïnstalleerd (moet worden geïnstalleerd op een
andere server dan de uPath enterprise software en de IMS) en een beeld met 20x-vergroting is verkregen met de juiste kleuring en
het juiste weefseltype, activeert de uPath enterprise software automatisch een volledige objectglaasjesanalyse (WSA).
6. WSA analyseert automatisch het volledige gescande beeld.
7. Als de WSA eenmaal is voltooid, wordt de patholoog binnen de uPath enterprise software op de hoogte gebracht dat de "analysis is
complete" (analyse is voltooid). De patholoog kan nu de specifieke ROI's selecteren die moeten worden gescoord. Een patholoog
kan een enkele ROI van een willekeurige grootte of meerdere ROI's selecteren. Als er meerdere ROI's worden geselecteerd, wordt
er een verzamelscore en een individuele score voor ieder ROI gegeven.
Kleuring
• Weefselpreparatie en -kleuring moeten worden uitgevoerd volgens de aanbevelingen in het methodeblad van de VENTANA PD-L1
(SP263) Assay.
• Alle nodige controles moeten worden uitgevoerd en de glaasjes moeten opnieuw worden gekleurd als de kleuring niet voldoet aan
de richtlijnen in het methodeblad van de VENTANA PD-L1 (SP263) Assay.
• De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme vereist het gebruik van de VENTANA PD-L1 (SP263)
Assay en eventuele aanvullende materialen of middelen die zijn vermeld in het methodeblad van de VENTANA PD-L1 (SP263) Assay
om weefsels voorafgaand aan analyse te kleuren. De VENTANA PD-L1 (SP263) Assay detecteert PD-L1-eiwit in FFPE NSCLC-
weefsel dat is gekleurd met de OptiView DAB IHC Detection Kit op een BenchMark ULTRA-instrument. Hoewel de VENTANA
PD-L1 (SP263) Assay PD-L1-eiwit detecteert in FFPE NSCLC-weefsel dat is gekleurd op een BenchMark ULTRA-instrument, is
de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme gevalideerd door middel van de OptiView DAB IHC
Detection Kit met kleuring op het BenchMark ULTRA-instrument.
Beeld vastleggen
De VENTANA DP 200-objectglaasjesscanner is vereist voor het scannen van de glaasjes. Beelden moeten worden gescand bij een
20x-vergroting. Als grote delen van het beeld onscherp zijn, wordt er aanbevolen de glaasjes opnieuw te scannen. Raadpleeg de
gebruikershandleiding van de VENTANA DP 200-objectglaasjesscanner (onderdeelnr. 1017149NL) voor meer informatie over scannen.
8 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Algemene navigatie: uPath enterprise software
De uPath enterprise software kan worden aangepast aan individuele en instellingsbehoeften, inclusief maar niet beperkt tot:
rapportconfiguratie en gebruikersinterface. Deze algoritmegids richt zich uitsluitend op de vereiste hulpmiddelen voor gebruik van de
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme. Raadpleeg de gebruikershandleiding van de uPath enterprise
software voor meer informatie over de uPath enterprise software.
Werkstroom van de patholoog
Een casus openen
Beelden van NSCLC-weefsel dat is gekleurd met de VENTANA PD-L1 (SP263) Assay kunnen worden geopend door te dubbelklikken
op een casus of door een casus te selecteren door in de uPath enterprise software op de viewer tab (het viewertabblad) te klikken
(Afbeelding 1).
Er wordt een scherm weergegeven met daarop alle beelden die aan een casus zijn gekoppeld (Afbeelding 2).
Afbeelding 2
Afbeelding 1
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 9

Als een objectglaasje dat is gekleurd met de VENTANA PD-L1 (SP263) Assay eenmaal is gescand op een VENTANA DP
200-objectglaasjesscanner bij 20x-vergroting, wordt het beeld geïmporteerd naar de uPath enterprise software en gekoppeld aan
een casus. De uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme activeert automatisch een WSA. De tijd die
het kost om de WSA-precomputingstap te voltooien, is afhankelijk van de serverspecificaties, de beeldgrootte en het aantal beelden
in de wachtrij. Als "waiting to start auto-analysis" (wachten tot automatische analyse wordt gestart) wordt weergegeven, wordt het
aantal beelden in de wachtrij dat nog moet worden geanalyseerd weergegeven. "Analyzing" (Analyseren) wordt weergegeven als de
WSA wordt uitgevoerd (Afbeelding 3 en 4). Als het beeld volledig is geanalyseerd door de WSA in de uPath enterprise software, wordt
"analysis successful" (analyse geslaagd) weergegeven onder het objectglaasjesbeeld in de viewer van de uPath enterprise software
(Afbeelding 5). Beelden kunnen niet worden gescoord voordat de WSA is voltooid.
ROI's van de gehele tumor tekenen: het tumorgebied selecteren
Gebruik de hulpmiddelknop Freehand (Vrij) in het vervolgkeuzemenu ROI (Afbeelding 6) om de tumorgebieden op het IHC-glaasjesbeeld te
selecteren die moeten worden geanalyseerd. Afbeelding 7 toont een beeld waarop een enkel ROI is getekend. Er kunnen aanvullende ROI's
worden getekend. Er verschijnt voor ieder geselecteerd gebied een ROI in het Slide Panel (Glaasjespaneel; Afbeelding 8).
Afbeelding 6 Afbeelding 7
Afbeelding 8
Afbeelding 5
Afbeelding 4
Afbeelding 3
10 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

ROI's van de gehele tumor tekenen: exclusiegebied
Bij het tekenen van ROI's is het uitsluiten van bepaalde gebieden mogelijk nodig; specifieke gebieden die moeten worden vermeden
of uitgesloten en voorbeelden hiervan worden weergegeven in de paragraaf over kleuringskenmerken hieronder. Gebruik het
exclusiehulpmiddel Freehand (Vrij) in het vervolgkeuzemenu Exclusion (Exclusie) (Afbeelding 9) om bepaalde specifieke gebieden uit te
sluiten (Afbeelding 10). Scan het glaasje opnieuw als grote delen van het beeld wazig of onscherp zijn.
Uitgesloten gebieden worden niet geanalyseerd door het algoritme en de gekleurde en ongekleurde TC binnen dit gebied worden
uitgesloten van het totale analysegebied. Als het ROI al is geanalyseerd en er een exclusie is gebruikt, moet het ROI opnieuw worden
geanalyseerd. De overlay en score worden navenant bijgewerkt.
Het tekenen van veel exclusiegebieden, zeker ingewikkelde Freehand (Vrije) exclusies, kan veel tijd in beslag nemen en van invloed
zijn op de efficiëntie van de werkstroom en slechts een kleine impact hebben op de uiteindelijke score. Als een casus een groot aantal
exclusies vereist, dient de patholoog het volgende te doen:
• Teken meerdere ROI's en sluit gedeeltes van het weefsel uit die als onscoorbaar worden aangemerkt met minimaal gebruik van het
hulpmiddel Exclusion (Exclusie).
• Beperk de exclusies en vervang de score handmatig door een andere score.
ROI's van de gehele tumor tekenen: verwijderen
Als het geselecteerde ROI van een gehele tumor niet optimaal is, kan het worden verwijderd. Selecteer het ROI van de gehele tumor door op
het midden van het ROI op het beeld te klikken en vervolgens op de knop Delete (Verwijderen) te klikken op het Slide Panel (Glaasjespaneel;
Afbeelding 11) of op het objectglaasjesbeeld nabij het ROI (Afbeelding 12). Er wordt een bevestigingsvenster weergegeven. Selecteer
Confirm (Bevestigen) om het geselecteerde ROI te verwijderen. Selecteer Cancel (Annuleren) om het ROI te bewaren.
Afbeelding 10Afbeelding 9
Afbeelding 11 Afbeelding 12
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 11

Het beeld is klaar om te worden geanalyseerd zodra er ROI's van de gehele tumor en/of exclusiegebieden zijn getekend. Selecteer het
ROI van de gehele tumor door op het midden van het ROI te klikken dat moet worden geanalyseerd of door op het ROI te klikken in
het Slide Panel (Glaasjespaneel). Selecteer voor ieder ROI de knop Image Analysis (Beeldanalyse) in het Slide Panel (Glaasjespaneel;
Afbeelding 13) of naast het ROI (Afbeelding 14).
Als de PD-L1-analyse is voltooid, worden de resultaten weergegeven in het Slide Panel (Glaasjespaneel) op twee plekken: onder Slide
Score (Glaasjesscore) en naast de ROI's (Afbeelding 15). De Slide Score (Glaasjesscore) is gebaseerd op een samenvatting van de
positiviteitsstatus van de tumorcellen van alle geselecteerde ROI's. Dit is ook de score die in het rapport verschijnt.
PD-L1-beeldanalyse
Afbeelding 15
Raadpleeg voor meer gedetailleerde informatie het uitvouwgedeelte in de Slide Score (Glaasjesscore) en het uitvouwgedeelte van
de ROI Details (ROI-details) door op het uitvouwpictogram te klikken (Afbeelding 16). Het uitvouwgedeelte van de Slide Score
(Glaasjesscore) wordt weergegeven (Afbeelding 17). Klik op het hetzelfde uitvouwpictogram om deze informatie weer te verbergen.
Afbeelding 17
Afbeelding 16
Afbeelding 13 Afbeelding 14
12 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

PD-L1-beeldanalyse: valse kleurenoverlay
Als de knop Analysis (Analyse) van de ROI's is ingedrukt en het weefsel is geanalyseerd, wordt er een kleurenoverlay weergegeven op het
ROI. Op het onderstaande beeld (Afbeelding 18) staan de rode cirkels voor cellen die zijn beoordeeld als positief gekleurd voor PD-L1 en
staan de blauwe cirkels voor cellen die zijn beoordeeld als negatief voor PD-L1. Als het beeld wordt opgepakt (klik met de linkermuisknop
en verplaats het beeld), verdwijnt de overlay (Afbeelding 19). De overlay verschijnt weer als de muisknop wordt losgelaten (Afbeelding 18).
Afbeelding 18
Afbeelding 19
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 13

Een glaasjesscore handmatig vervangen
Scores kunnen handmatig worden vervangen door op het uitvouwpictogram van de Slide Score (Glaasjesscore) te klikken in het Slide Panel
(Glaasjespaneel) naast de Slide Score (Glaasjesscore) (Afbeelding 16). Het uitvouwgedeelte van de Slide Score (Glaasjesscore) wordt
weergegeven (Afbeelding 20). Door de knop Edit (Bewerken) (Afbeelding 20) te selecteren op het uitvouwgedeelte van de Slide Score
(Glaasjesscore) kan de gebruiker handmatig een score typen (Afbeelding 21). In het veld Comments (Opmerkingen) kunnen opmerkingen
worden ingevoerd over de casus en/of de beslissing om de geautomatiseerde score te vervangen. Voor PD-L1 kunnen scores van 0-100%
handmatig worden ingevoerd. Selecteer de optie Confirm (Bevestigen) na het invoeren van een handmatige score (Afbeelding 22).
Er wordt een bevestigingsmelding weergegeven; selecteer "Yes" (Ja).
De score op het Slide Panel (Glaasjespaneel) geeft nu de handmatig vervangen score weer. De beeldanalysescore naast de ROI's is niet
langer aanwezig (Afbeelding 23). De gebruiker kan het beeld opnieuw analyseren door op de staafgrafiekknop te klikken (Afbeelding 13,
Afbeelding 14).
Afbeelding 21 Afbeelding 22
Afbeelding 20
Afbeelding 23
14 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Raadpleeg het methodeblad en de interpretatiegids van de VENTANA PD-L1 (SP263) Assay.
PD-L1-evaluatie
NSCLC neoplastische cellen die zijn gelabeld met de VENTANA PD-L1 (SP263) Assay worden geëvalueerd op het positiviteitspercentage
van de TC met membraankleuring van elke intensiteit. De immunohistochemische kleuring bij NSCLC is membraneus en/of
cytoplasmisch en kan homogeen of heterogeen worden uitgedrukt in het neoplasma. Er wordt geen rekening gehouden met
cytoplasmatische kleuring van tumorcellen in de TC-positiviteitsscore. Membraankleuring kan een discontinu, omringend of
basolateraal patroon hebben. Een negatief controleantilichaam met hetzelfde isotype wordt gebruikt om de aanwezigheid van
achtergrond in testmonsters te evalueren.
Kleuringsevaluatie van het PD-L1-algoritme
De patholoog die de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme gebruikt, dient bekend te zijn met het
handmatig scoren van de VENTANA PD-L1 (SP263) Assay. De patholoog moet het hulpmiddel Freehand (Vrij) gebruiken om het
volledige tumorgebied te omcirkelen. De patholoog moet refereren naar het bijbehorende H&E- en negatief controleglaasje alvorens
de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme te gebruiken. Houd bij het selecteren van de gebieden voor
analyse rekening met de beperkingen die zijn beschreven in de paragrafen Beperkingen en Gebieden om te vermijden. Als de patholoog
het niet eens is met de score die door de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme is bepaald, dient de
patholoog deze handmatig te vervangen.
Niet-evalueerbare casussen omvatten, maar zijn niet beperkt tot, casussen met onvoldoende geschikt tumormateriaal, onacceptabele
morfologie en storende achtergrond. NSCLC-casussen met voldoende geschikte TC (zoals bepaalde door de scorende patholoog) en
geen storende achtergrond op het PD-L1 IHC-glaasje zijn acceptabel voor evaluatie.
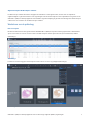
Kleuringskenmerken
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 15

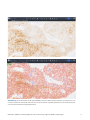
Afbeelding 24: gescande beelden van IHC-gekleurd NSCLC-weefsel in uPath enterprise software; voor analyse (boven) en
na analyse (onder). De rode overlay staat voor cellen die zijn beoordeeld als positief gekleurde TC en de blauwe overlay voor
cellen die zijn beoordeeld als negatief gekleurde TC.
De volgende afbeeldingen zijn beelden met verschillende kleuringspatronen en PD-L1-expressieniveau's (24-25). PD-L1-expressie in TC
wordt uitgedrukt als een percentage (0-100).
16 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

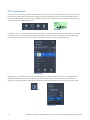
Afbeelding 25: gescande beelden van IHC-gekleurd NSCLC-weefsel in uPath enterprise software; voor analyse (boven) en
na analyse (onder). De rode overlay staat voor cellen die zijn beoordeeld als positief gekleurde TC en de blauwe overlay voor
cellen die zijn beoordeeld als negatief gekleurde TC.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 17

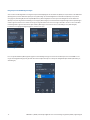
Afbeelding 26: voorbeeld van gemiste tumorcelidentificatie. Gescande beelden van IHC-gekleurd NSCLC-weefsel in uPath
enterprise software; voor analyse (boven) en na analyse (onder). De rode overlay staat voor cellen die zijn beoordeeld als
positief gekleurde tumorcellen en de blauwe overlay voor cellen die zijn beoordeeld als negatief gekleurde tumorcellen.
Voorbeelden van gemiste tumorcellen
De volgende afbeeldingen tonen verschillende voorbeelden van gemiste tumorcellen (26-28).
18 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Afbeelding 27: voorbeeld van gemiste tumorcelidentificatie. Gescande beelden van IHC-gekleurd NSCLC-weefsel in uPath
enterprise software; voor analyse (boven) en na analyse (onder). De rode overlay staat voor cellen die zijn beoordeeld als
positief gekleurde tumorcellen en de blauwe overlay voor cellen die zijn beoordeeld als negatief gekleurde tumorcellen.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 19

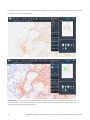
Afbeelding 28: voorbeeld van gemiste tumorcelidentificatie. Gescande beelden van IHC-gekleurd NSCLC-weefsel in uPath
enterprise software; voor analyse (boven) en na analyse (onder). De rode overlay staat voor cellen die zijn beoordeeld als
positief gekleurde tumorcellen en de blauwe overlay voor cellen die zijn beoordeeld als negatief gekleurde tumorcellen.
20 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Gebieden om te vermijden bij gebruik van het algoritme
Zoals eerder aangegeven moeten tijdens het tekenen van het ROI bepaalde gebieden worden uitgesloten van het gehele tumorgebied
met behulp van het hulpmiddel Exclusion (Exclusie) of worden vermeden bij het definiëren van een tumorgebied met een ROI. Bij
gebruik van de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme moeten pathologen het scoren van gebieden
vermijden als deze gebieden normaal gesproken worden vermeden bij handmatig scoren. De algehele score voor PD-L1 kan worden
vervangen als de gebruiker het niet eens is met de score van de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme.
Afbeelding 29: voorbeeld van necrotisch weefsel voor analyse (boven) en na analyse (onder). De gebruiker dient zich
te houden aan de richtlijnen in het methodeblad van de VENTANA PD-L1 (SP263) Assay. Het is belangrijk om necrotisch
weefsel uit te sluiten, omdat het de manier waarop het algoritme weefselmonsters scoort kan beïnvloeden en kan bijdragen
aan een onnauwkeurige PD-L1 TC-positiviteitsscore.
Voorbeeld van necrose
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 21

Voorbeeld van gevouwen weefsel
Afbeelding 30: voorbeeld van gevouwen weefsel. De gebruiker dient zich te houden aan de richtlijnen in het methodeblad
van de VENTANA PD-L1 (SP263) Assay. Het is belangrijk om gevouwen weefsel uit te sluiten, omdat het de manier waarop het
algoritme weefselmonsters scoort kan beïnvloeden en kan leiden tot een onnauwkeurige PD-L1 TC-positiviteitsscore.
22 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Voorbeeld van achtergrondkleuring
Afbeelding 31: voorbeeld van achtergrondkleuring voor analyse (boven) en na analyse (onder). De gebruiker dient zich te
houden aan de richtlijnen in het methodeblad van de VENTANA PD-L1 (SP263) Assay. Het is belangrijk om achtergrondkleuring
uit te sluiten, omdat het de manier waarop het algoritme weefselmonsters scoort kan beïnvloeden en kan bijdragen aan een
onnauwkeurige PD-L1 TC-positiviteitsscore.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 23

Voorbeeld van vuil
Afbeelding 32: voorbeeld van vuil voor analyse (boven) en na analyse (onder). De gebruiker dient zich te houden aan
de richtlijnen in het methodeblad van de VENTANA PD-L1 (SP263) Assay. Het is belangrijk om vuil uit te sluiten, omdat het
de manier waarop het algoritme weefselmonsters scoort kan beïnvloeden en kan bijdragen aan een onnauwkeurige PD-L1
TC-positiviteitsscore.
24 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Voorbeeld van macrofagen
Afbeelding 33: voorbeeld van macrofagen voor analyse (boven) en na analyse (onder). De gebruiker dient zich te houden
aan de richtlijnen in het methodeblad van de VENTANA PD-L1 (SP263) Assay. Het is belangrijk om macrofagen uit te sluiten,
omdat deze de manier waarop het algoritme weefselmonsters scoort kunnen beïnvloeden en kunnen bijdragen aan een
onnauwkeurige PD-L1 TC-positiviteitsscore.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 25

De prestatie van de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme werd geëvalueerd aan de hand van
onderzoeken naar inter-readerovereenkomst, intra-readerovereenkomst, methodevergelijking, inter-scannerovereenkomst en
intra-scannerovereenkomst. Alle analyses werden uitgevoerd met de uPath PD-L1-beeldanalyseapplicatie voor het NSCLC-algoritme
op de uPath enterprise software. Deelnemende pathologen volgden gedurende de onderzoeken de richtlijnen in deze algoritmegids.
Alle weefselmonsters werden gekleurd met de VENTANA PD-L1 (SP263) Assay met behulp van de OptiView DAB IHC Detection Kit op
een BenchMark ULTRA-instrument en ze werden gescand met de VENTANA DP 200-objectglaasjesscanner bij een 20x-vergroting.
Er werden alleen objectglaasjes of glaasjesbeelden gebruikt die acceptabel waren volgens de richtlijnen in het methodeblad van de
VENTANA PD-L1 (SP263) Assay. Alles onder de grens werd beoordeeld als negatief en alles gelijk aan of groter dan de grens van 50%
werd beoordeeld als positief.
Prestatiekenmerken
Vergelijking van methoden
Het PD-L1 (SP263)-methodevergelijkingsonderzoek voor de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme
en de handmatige interpretatie (microscoop) bestonden uit 180 gedeïdentificeerde gearchiveerde NSCLC-secties. De testmonsters van
het onderzoek hadden scores voor positieve kleuring die uiteenliepen van 0 tot 100%, en werden door drie verschillende pathologen
geïnterpreteerd.
Tabel 1. Overeenstemming: handmatige interpretatie vs. beeldanalyse (grenswaarde 50%)
MR-resultaat Overeenstemming
Beoordelaar IA-resultaat Positief Negatief Totaal Meting [a] % (n/N) 95% CI [b]
Totaal
Positief 174 25 199 PPA(%) 96,7 (174/180) (94,0; 98,9)
Negatief 6 335 341 NPA(%) 93,1 (335/360) (90,2; 95,6)
Totaal
180 360 540 OPA(%) 94,3 (509/540) (92,0; 96,1)
Beoordelaar 1
Positief 59 11 70 PPA(%) 98,3 (59/60) (91,1; 99,7)
Negatief 1 109 110 NPA(%) 90,8 (109/120) (84,3; 94,8)
Totaal 60 120 180 OPA(%) 93,3 (168/180) (88,7; 96,1)
Beoordelaar 2
Positief
56 9 65 PPA(%) 93,3 (56/60) (84,1; 97,4)
Negatief 4 111 115 NPA(%) 92,5 (111/120) (86,4; 96,0)
Totaal 60 120 180 OPA(%) 92,8 (167/180) (88,0; 95,7)
Beoordelaar 3
Positief 59 5 64 PPA(%) 98,3 (59/60) (91,1; 99,7)
Negatief 1 115 116 NPA(%) 95,8 (115/120) (90,6; 98,2)
Totaal 60 120 180 OPA(%) 96,7 (174/180) (92,9; 98,5)
MR=handmatige interpretatie, IA=beeldanalyse
[a] PPA = positieve procentuele overeenstemming; NPA = negatieve procentuele overeenstemming; OPA = totale procentuele overeenstemming.
[b] Voor de totale overeenstemming werden de betrouwbaarheidsintervallen berekend aan de hand van de percentuele bootstrapmethode.
De betrouwbaarheidsintervallen voor het resultaat van iedere beoordelaar werden berekend aan de hand van de Wilson-scoringsmethode.
Opmerking: alleen observaties met niet-ontbrekende evaluaties werden opgenomen in deze analyse.
Opmerking: observaties die in de uiteindelijke klinische scorebin van <1% of >=1% tot <50% vielen, waren negatief. Opmerking: observaties die in
de uiteindelijke klinische scorebin van >=<50% vielen, waren positief.
26 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Modaal resultaat Overeenstemming
Methode Lezen Beoordelaarsresultaat Positief Negatief Totaal Meting [a] % (n/N) 95% CI [b]
WTA IA
Totaal
Positief 62 0 62 PPA 98,4 (62/63) (95,7; 100,0)
Negatief 1 116 117 NPA 100,0 (116/116) (96,8; 100,0)
Totaal 63 116 179 OPA 99,4 (178/179) (98,3; 100,0)
Lezen 1
Positief 21 0 21 PPA 100,0 (21/21) (84,5; 100,0)
Negatief 0 38 38 NPA 100,0 (38/38) (90,8; 100,0)
Totaal 21 38 59 OPA 100,0 (59/59) (93,9; 100,0)
Lezen 2
Positief
20 0 20 PPA 95,2 (20/21) (77,3; 99,2)
Negatief 1 39 40 NPA 100,0 (39/39) (91,0; 100,0)
Totaal 21 39 60 OPA 98,3 (59/60) (91,1; 99,7)
Lezen 3
Positief 21 0 21 PPA 100,0 (21/21) (84,5; 100,0)
Negatief 0 39 39 NPA 100,0 (39/39) (91,0; 100,0)
Totaal 21 39 60 OPA 100,0 (60/60) (94,0; 100,0)
Tabel 3. Overeenstemming tussen dezelfde beoordelaar van de PD-L1-status (50%)
IA=beeldanalyse
[a] PPA = positieve procentuele overeenstemming; NPA = negatieve procentuele overeenstemming; OPA = totale procentuele overeenstemming.
[b] Voor de totale overeenstemming werden de betrouwbaarheidsintervallen berekend aan de hand van de percentuele bootstrapmethode, gestratificeerd door
screeningbins. De betrouwbaarheidsintervallen voor het resultaat van iedere ronde werden berekend aan de hand van de Wilson-scoringsmethode.
Opmerking: alleen observaties met niet-ontbrekende evaluaties werden opgenomen in deze analyse.
Opmerking: observaties die in de uiteindelijke klinische scorebin van <1% of >=1% tot <50% vielen, waren negatief. Opmerking: observaties die in de uiteindelijke
klinische scorebin van >=<50% vielen, waren positief.
Opmerking: observatie met het glaasje ID C103 werd uitgesloten van deze analyse vanwege onverenigbare gegevensproblemen.
Reproduceerbaarheidsonderzoeken voor pathologen
De reproduceerbaarheidsonderzoeken voor pathologen voor de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme
bestonden uit 180 gedeïdentificeerde gearchiveerde NSCLC-secties voor inter-readerbeoordeling door verschillende beoordelaars en
60 gedeïdentificeerde gearchiveerde NSCLC-secties. De testmonsters van het onderzoek hadden scores voor positieve kleuring die
uiteenliepen van 0-100%, en werden door drie verschillende pathologen geïnterpreteerd.
Modaal resultaat Overeenstemming
Methode Beoordelaar Beoordelaarsresultaat Positief Negatief Totaal Meting [a] % (n/N) 95% CI [b]
WTA IA
Totaal
Positief 194 5 199 PPA 95,1 (194/204) (92,5; 97,5)
Negatief 10 331 341 NPA 98,5 (331/336) (97,1; 99,7)
Totaal 204 336 540 OPA 97,2 (525/540) (95,9; 98,5)
Beoordelaar 1
Positief 68 2 70 PPA 100,0 (68/68) (94,7; 100,0)
Negatief 0 110 110 NPA 98,2 (110/112) (93,7; 99,5)
Totaal 68 112 180 OPA 98,9 (178/180) (96,0; 99,7)
Beoordelaar 2
Positief
64 1 65 PPA 94,1 (64/68) (85,8; 97,7)
Negatief 4 111 115 NPA 99,1 (111/112) (95,1; 99,8)
Totaal 68 112 180 OPA 97,2 (175/180) (93,7; 98,8)
Beoordelaar 3
Positief 62 2 64 PPA 91,2 (62/68) (82,1; 95,9)
Negatief 6 110 116 NPA 98,2 (110/112) (93,7; 99,5)
Totaal 68 112 180 OPA 95,6 (172/180) (91,5; 97,7)
IA=beeldanalyse
[a] PPA = positieve procentuele overeenstemming; NPA = negatieve procentuele overeenstemming; OPA = totale procentuele overeenstemming.
[b] Voor de totale overeenstemming werden de betrouwbaarheidsintervallen berekend aan de hand van de percentuele bootstrapmethode. De
betrouwbaarheidsintervallen voor het resultaat van iedere beoordelaar werden berekend aan de hand van de Wilson-scoringsmethode.
Opmerking: alleen observaties met niet-ontbrekende evaluaties werden opgenomen in deze analyse.
Opmerking: observaties die in de uiteindelijke klinische scorebin van <1% of >=1% tot <50% vielen, waren negatief. Opmerking: observaties die in de uiteindelijke
klinische scorebin van >=<50% vielen, waren positief.
Tabel 2. Inter-readerovereenkomst van de PD-L1-status (50%)
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 27

Modaal resultaat Overeenstemming
Methode Scanner Scannerresultaat Positief Negatief Totaal Meting [a] % (n/N) 95% CI [b]
WTA IA
Totaal
Positief 204 2 206 PPA 100,0 (204/204) (98,2; 100,0)
Negatief
0 334 334 NPA 99,4 (334/336) (98,5; 100,0)
Totaal 204 336 540 OPA 99,6 (538/540) (99,1; 100,0)
Scanner 1
Positief 72 1 73 PPA 100,0 (72/72) (94,9; 100,0)
Negatief 0 107 107 NPA 99,1 (107/108) (97,1; 100,0)
Totaal 72 108 180 OPA 99,4 (179/180) (98,3; 100,0)
Scanner 2
Positief
69 1 70 PPA 100,0 (69/69) (94,7; 100,0)
Negatief 0 110 110 NPA 99,1 (110/111) (97,5; 100,0)
Totaal 69 111 180 OPA 99,4 (179/180) (98,3; 100,0)
Scanner 3
Positief 63 0 63 PPA 100,0 (63/63) (94,3; 100,0)
Negatief 0 117 117 NPA 100,0 (117/117) (96,8; 100,0)
Totaal 63 117 180 OPA 100,0 (180/180) (97,9; 100,0)
Tabel 5. Overeenstemming tussen dezelfde scanner van de PD-L1-status (50%)
IA=beeldanalyse
[a] PPA = positieve procentuele overeenstemming; NPA = negatieve procentuele overeenstemming; OPA = totale procentuele overeenstemming.
[b] De betrouwbaarheidsintervallen werden berekend aan de hand van de percentuele bootstrapmethode.
Opmerking: alleen observaties met niet-ontbrekende evaluaties werden opgenomen in deze analyse.
Opmerking: observaties die in de klinische scorebin van <1% of >=1% tot <50% vielen, waren negatief. Observaties die in de uiteindelijke klinische scorebin van >=<50%
vielen, waren positief.
Reproduceerbaarheidsonderzoeken voor scanners
De reproduceerbaarheidsonderzoeken voor scanners voor de uPath PD-L1 (SP263)-beeldanalyseapplicatie voor het NSCLC-algoritme
bestonden uit 60 gedeïdentificeerde gearchiveerde NSCLC-secties. De testmonsters van het onderzoek hadden scores voor positieve
kleuring die uiteenliepen van 0-100%. De monsters werden gescand door een getrainde gebruiker voor 3 scans op elk van de 3
verschillende VENTANA DP 200-objectglaasjesscanners bij een 20x-vergroting en ze werden geanalyseerd met de uPath PD-L1 (SP263)-
beeldanalyseapplicatie voor het NSCLC-algoritme. De scores werden vergeleken voor de verschillende scanners.
Modaal resultaat Overeenstemming
Methode Scanner Beoordelaarsresultaat Positief Negatief Totaal Meting [a] % (n/N) 95% CI [b]
WTA IA
Totaal
Positief 201 5 206 PPA 97,1 (201/207) (95,2; 100,0)
Negatief 6 328 334 NPA 98,5 (328/333) (96,4; 100,0)
Totaal 207 333 540 OPA 98,0 (529/540) (96,5; 99,4)
Scanner 1
Positief 69 4 73 PPA 100,0 (69/69) (94,7; 100,0)
Negatief 0 107 107 NPA 96,4 (107/111) (91,7; 100,0)
Totaal 69 111 180 OPA 97,8 (176/180) (94,4; 100,0)
Scanner 2
Positief
69 1 70 PPA 100,0 (69/69) (94,7; 100,0)
Negatief 0 110 110 NPA 99,1 (110/111) (97,5; 100,0)
Totaal 69 111 180 OPA 99,4 (179/180) (98,3; 100,0)
Scanner 3
Positief 63 0 63 PPA 91,3 (63/69) (85,7; 100,0)
Negatief 6 111 117 NPA 100,0 (111/111) (96,7; 100,0)
Totaal 69 111 180 OPA 96,7 (174/180) (95,0; 100,0)
IA=beeldanalyse
[a] PPA = positieve procentuele overeenstemming; NPA = negatieve procentuele overeenstemming; OPA = totale procentuele overeenstemming.
[b] De betrouwbaarheidsintervallen werden berekend aan de hand van de percentuele bootstrapmethode.
Opmerking: alleen observaties met niet-ontbrekende evaluaties werden opgenomen in deze analyse.
Opmerking: observaties die in de klinische scorebin van <1% of >=1% tot <50% vielen, waren negatief. Observaties die in de uiteindelijke klinische scorebin van >=<50%
vielen, waren positief.
Tabel 4. Overeenstemming tussen verschillende scanners van de PD-L1-status (50%)
28 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Problemen oplossen
Een mislukte analyse opnieuw starten
Als er zich een fout voordoet tijdens het uitvoeren van een WSA in de uPath enterprise software, wordt "analysis error" (analysefout)
weergegeven in een rode balk onder het glaasjesbeeld (Afbeelding 34).
Meld u aan als beheerder om de analyse in de uPath enterprise software opnieuw te starten (Afbeelding 35).
Klik in de Administrator Settings (Beheerdersinstellingen) op Job Queue (Takenwachtrij; Afbeelding 36).
Afbeelding 34
Afbeelding 35
Afbeelding 36
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 29

Selecteer het tabblad Failed (Mislukt) om alle glaasjes met een mislukte analyse weer te geven. Selecteer het glaasje dat moet worden
geanalyseerd om de knoppen Cancel Job (Taak annuleren) en Start Job (Taak starten) te activeren (Afbeelding 37).
Afbeelding 37
Afbeelding 38
Selecteer Start Job (Taak starten) om de analyse te starten. Indien succesvol wordt "Job scheduled succesfully" (Taak is gepland)
weergegeven in een groene balk bovenaan het scherm (Afbeelding 38).
30 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Het glaasjesbeeld wordt nu weergegeven op de pagina Pending (In afwachting). Eenmaal op deze pagina is het mogelijk om taken te
herschikken op volgorde van prioriteit als de wachtrij meerdere beelden bevat (Afbeeldingen 39).
Na een aantal seconde wordt het bovenste glaasjesbeeld verplaatst naar de pagina In-Progress (In behandeling) en wordt er een
voortgangsbalk voor weergegeven. Het is niet nodig om op deze pagina te blijven om de analyse uit te voeren (Afbeelding 40).
Afbeelding 40
Afbeelding 39
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 31

Als een glaasjesanalyse is voltooid, wordt het beeld verplaatst naar de pagina Canceled/Complete (Geannuleerd/voltooid) (Afbeelding 41).
In de uPath-viewer wordt nu "analysis succesful" (analyse geslaagd) weergegeven onder het glaasjesbeeld en alle analytische
functionaliteit voor het glaasje is beschikbaar (Afbeelding 42).
Afbeelding 42
Afbeelding 41
32 uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids

Bronvermeldingen
1. Ferlay J, Soerjomataram I, Ervik M, et al. GLOBOCAN 2012 v1.0. Cancer Incidence and Mortality Worldwide: IARC
CancerBase No. 11. http://globocan.iarc.fr. Published 2013-12-12. Updated 2014-01-09. Accessed 2016-02-08.
2. Howlader N, Noone AM, Krapcho M, et al. (eds). SEER Cancer Statistics Review (CSR),1975-2012. National Cancer
Institute. http://seer.cancer.gov/csr/1975_2012/. Published 2015-04-23. Updated 2015-11-18. Accessed 2016-02-08.
3. Keir ME, Butte MJ, Freeman GJ, et al. PD-1 and its ligands in tolerance and immunity. Annu Rev Immunol 2008;26:677-704.
4. Blank C, Mackensen A. Contribution of the PD-L1/PD-1 pathway to T-cell exhaustion: an update on implications
for chronic infections and tumor evasion. Cancer Immunol Immunother. 2007;56(5):739-745.
5. Butte MJ, Keir ME, Phamduy TB, et al. Programmed death-1 ligand 1 interacts specifically with the
B7-1 costimulatory molecule to inhibit T-cell responses. Immunity. 2007;27(1):111-122.
6. Dong H, Zhu G, Tamada K, et al. B7-H1, a third member of the B7 family, co-stimulates T-cell
proliferation and interleukin-10 secretion. Nat Med. 1999;5(12):1365-1369.
7. Massard C, Gordon MS, Sharma S, et al. Safety and efficacy of durvalumab (MEDI4736), an anti-programmed cell death ligand-1
immune checkpoint inhibitor, in patients with advanced urothelial bladder cancer. J Clin Oncol. 2016;34(26):3119-3126.
uPath PD-L1 (SP263)-beeldanalyseapplicatie voor niet-kleincellige longkanker (NSCLC) algoritmegids 33

Roche Diagnostics GmbH
Sandhofer Strasse 116
D-68305 Mannheim
Germany
www.roche.com
www.ventana.com
© 2020 Ventana Medical Systems, Inc. en Roche Diagnostics
International, Inc. Alle rechten voorbehouden.
VENTANA, BENCHMARK, OPTIVIEW, UPATH en het VENTANA-
logo zijn handelsmerken van Roche. Alle andere handelsmerken
zijn eigendom van hun respectievelijke eigenaren.
1019285NL Rev A
2020-03-31
08764174001
-
 1
1
-
 2
2
-
 3
3
-
 4
4
-
 5
5
-
 6
6
-
 7
7
-
 8
8
-
 9
9
-
 10
10
-
 11
11
-
 12
12
-
 13
13
-
 14
14
-
 15
15
-
 16
16
-
 17
17
-
 18
18
-
 19
19
-
 20
20
-
 21
21
-
 22
22
-
 23
23
-
 24
24
-
 25
25
-
 26
26
-
 27
27
-
 28
28
-
 29
29
-
 30
30
-
 31
31
-
 32
32
-
 33
33
-
 34
34
-
 35
35
-
 36
36
-
 37
37
-
 38
38
Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Handleiding
- Type
- Handleiding
Gerelateerde papieren
-
Roche BenchMark ULTRA Interpretation Guide
-
Roche BenchMark ULTRA Handleiding
-
Roche uPath IVD Handleiding
-
Roche BenchMark XT/LT Handleiding
-
Roche BenchMark ULTRA Handleiding
-
Roche BenchMark GX Handleiding
-
Roche Virtuoso Handleiding
-
Roche VENTANA DP 200 Handleiding
-
Roche Virtuoso Handleiding
-
Roche iScan HT IVD Handleiding










































